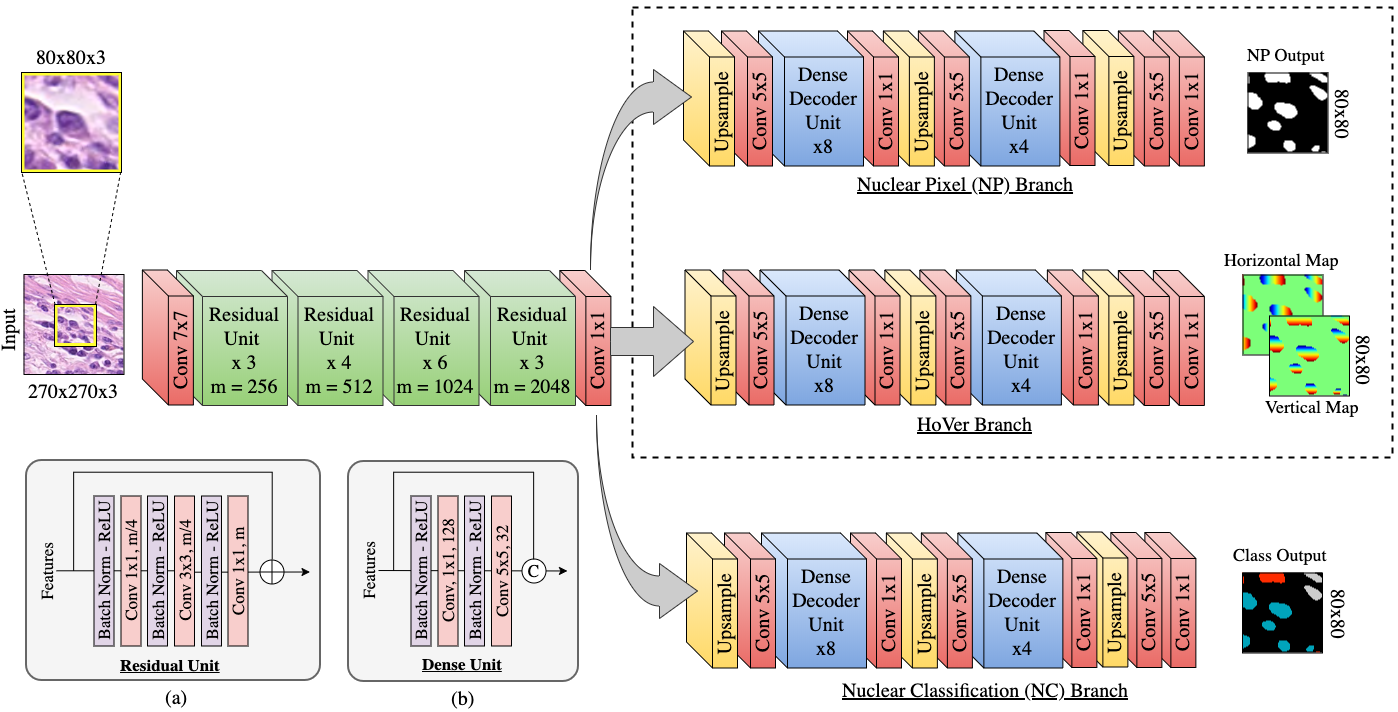
HoVer-Net: 多组织病理图像中核的同步分割与分类
一个多分支网络,可在单个网络中同时执行细胞核实例分割和分类。该网络利用核像素到其质心的水平和垂直距离来分离聚集的细胞。专用的上采样分支用于对每个分割实例的核类型进行分类。
医学图像分析论文链接。
这是HoVer-Net的官方PyTorch实现。原始TensorFlow版本的代码请参考此分支。该存储库可用于训练HoVer-Net并处理图像块或全幻灯片图像。作为此存储库的一部分,我们提供了在以下数据集上训练的模型权重:
检查点的链接可以在下面的推理描述中找到。
设置环境
conda env create -f environment.yml
conda activate hovernet
pip install torch==1.6.0 torchvision==0.7.0
上面我们安装了PyTorch 1.6版本和CUDA 10.2。
存储库结构
以下是存储库中的主要目录:
dataloader/: 数据加载器和增强管道docs/: 存储库中使用的图片/GIFmetrics/: 指标计算脚本misc/: 实用工具models/: 模型定义,以及主要运行步骤和超参数设置run_utils/: 定义训练/验证循环和回调
以下是存储库中的主要可执行脚本:
config.py: 配置文件dataset.py: 定义数据集类extract_patches.py: 从原始图像中提取patchescompute_stats.py: 主要指标计算脚本run_train.py: 主要训练脚本run_infer.py: 用于图块和WSI处理的主要推理脚本convert_chkpt_tf2pytorch: 将原始存储库中训练的tensorflow.npz模型转换为pytorch支持的.tar格式。
运行代码
训练
数据格式
对于训练,必须使用extract_patches.py提取patches。对于实例分割,patches存储为4维numpy数组,通道为[RGB, inst]。这里,inst是实例分割的ground truth。即像素范围从0到N,其中0是背景,N是该特定图像的细胞核实例数量。
对于同时进行实例分割和分类,patches存储为5维numpy数组,通道为[RGB, inst, type]。这里,type是细胞核类型的ground truth。即每个像素范围从0-K,其中0是背景,K是类别数。
在训练之前:
- 在
config.py中设置数据目录的路径 - 在
config.py中设置检查点保存路径 - 在
models/hovernet/opt.py中设置预训练的Preact-ResNet50权重路径。在这里下载权重。 - 在
models/hovernet/opt.py中修改超参数,包括epoch数和学习率。
用法和选项
用法:
python run_train.py [--gpu=<id>] [--view=<dset>]
python run_train.py (-h | --help)
python run_train.py --version
选项:
-h --help 显示此字符串。
--version 显示版本。
--gpu=<id> 逗号分隔的GPU列表。
--view=<dset> 在增强后可视化图像。选择'train'或'valid'。
示例:
要在训练前可视化训练数据集作为健全性检查,请使用:
python run_train.py --view='train'
要使用GPU 0和1初始化训练脚本,命令是:
python run_train.py --gpu='0,1'
推理
数据格式
输入:
- 标准图像文件,包括
png、jpg和tiff。 - OpenSlide支持的WSI,包括
svs、tif、ndpi和mrxs。
输出:
- 图像块和全幻灯片图像都输出一个
json文件,包含以下键:- 'bbox': 每个细胞核的边界框坐标
- 'centroid': 每个细胞核的质心坐标
- 'contour': 每个细胞核的轮廓坐标
- 'type_prob': 每个细胞核的每类概率(默认配置不输出)
- 'type': 每个细胞核的类别预测
- 图像块输出一个
mat文件,包含以下键:- 'raw': 网络的原始输出(默认配置不输出)
- 'inst_map': 包含0到N的值的实例图,其中N是细胞核数量
- 'inst_type': 长度为N的列表,包含每个细胞核的预测
- 图像块输出一个
png文件,显示原始RGB图像上的细胞核边界叠加
模型权重
可以提供按照上述说明训练HoVer-Net得到的模型权重来处理输入图像/WSI。或者,可以使用以下任何预训练模型权重来处理数据。这些检查点最初使用TensorFlow训练,并使用convert_chkpt_tf2pytorch.py进行转换。提供的检查点要么只训练用于分割,要么用于同时分割和分�类。注意,我们不提供CPM17和Kumar的分割和分类模型,因为没有分类标签。
重要: CoNSeP、Kumar和CPM17检查点使用原始模型模式,而PanNuke和MoNuSAC使用快速模型模式。有关更多信息,请参阅下面的推理说明。
分割和分类:
仅分割:
访问完整的检查点目录及文件名描述README,请点击这里。
如果使用上述任何检查点,请确保引用相应的论文。
用法和选项
用法:
run_infer.py [选项] [--help] <命令> [<参数>...]
run_infer.py --version
run_infer.py (-h | --help)
选项:
-h --help 显示此字符串。
--version 显示版本。
--gpu=<id> GPU列表。[默认: 0]
--nr_types=<n> 要预测的细胞核类型数量。[默认: 0]
--type_info_path=<path> 定义类型id、类型名称和预期叠加颜色之间映射的json路径。[默认: '']
--model_path=<path> 已保存检查点的路径。
--model_mode=<mode> ��原始HoVer-Net或PanNuke / MoNuSAC中使用的简化版本,'original'或'fast'。[默认: fast]
--nr_inference_workers=<n> 推理期间的工作进程数。[默认: 8]
--nr_post_proc_workers=<n> 后处理期间的工作进程数。[默认: 16]
--batch_size=<n> 批量大小。[默认: 128]
图块处理选项:
--input_dir=<path> 输入数据目录的路径。假设文件不嵌套在目录中。
--output_dir=<path> 输出目录的路径。
--draw_dot 在叠加层上绘制细胞核质心。[默认: False]
--save_qupath 可选择输出与QuPath v0.2.3兼容的格式。[默认: False]
--save_raw_map 是否保存原始预测。[默认: False]
WSI处理选项:
--input_dir=<path> 输入数据目录的路径。假设文件不嵌套在目录中。
--output_dir=<path> 输出目录的路径。
--cache_path=<path> 缓存路径。应放置在至少100GB的SSD上。[默认: cache]
--mask_dir=<path> 包含组织掩码的目录路径。
应与相应的WSI具有相同的名称。[默认: '']
--proc_mag=<n> 用于WSI处理的放大倍数(物镜倍率)。[默认: 40]
--ambiguous_size=<int> 定义沿分割网格的模糊区域以进行重新后处理。[默认: 128]
--chunk_shape=<n> 处理的块形状。[默认: 10000]
--tile_shape=<n> 处理的图块形状。[默认: 2048]
--save_thumb 保存缩略图。[默认: False]
--save_mask 保存掩码。[默认: False]
上述命令可以从命令行使用或通过可执行脚本使用。我们提供了两个示例可执行脚本:一个用于图块处理,一个用于WSI处理。要运行这些脚本,首先使用chmod +x run_tile.sh和chmod +x run_tile.sh使它们可执行�。然后使用./run_tile.sh和./run_wsi.sh运行。
中间结果存储在缓存中。因此,请确保指定的缓存位置有足够的空间!最好确保缓存位置是SSD。
请注意,在运行推理时选择正确的模型模式很重要。"原始"模型模式指的是原始医学图像分析论文中描述的方法,使用270x270补丁输入和80x80补丁输出。"快速"模型模式使用256x256补丁输入和164x164补丁输出。在Kumar、CPM17和CoNSeP上训练的模型检查点来自我们的原始发表,因此必须使用"原始"模式。对于PanNuke和MoNuSAC,必须选择"快速"模式。我们提供的每个检查点的模型模式都在文件名中给出。此外,如果使用仅针对分割训练的模型,必须将nr_types设置为0。
type_info.json用于指定叠加层中使用的RGB颜色。请确保为不同的数据集修改此文件,并且如果您想要通常控制叠加边界颜色。
作为我们瓦片处理实现的一部分,我们添加了一个选项,以兼容QuPath的形式保存输出。
请查看examples/usage.ipynb了解如何使用输出。
叠加分割和分类预测
<p float="left"> <img src="https://yellow-cdn.veclightyear.com/835a84d5/35f1430c-4704-419e-abb1-bd922cc4b210.gif" alt="Segmentation" width="870" /> </p>在CoNSeP数据集上训练的HoVer-Net的叠加结果。细胞核边界的颜色表示细胞核的类型。<br /> 蓝色:上皮<br /> 红色:炎症<br /> 绿色:纺锤形<br /> 青色:其他
数据集
从此链接下载我们论文中使用的CoNSeP数据集。<br /> 从此链接下载Kumar、CPM-15、CPM-17和TNBC数据集。<br /> 下载
真实标注文件采用.mat格式,有关更多信息,请参阅数据集附带的README。
与原始TensorFlow实现的比较
以下我们报告了使用此存储库(PyTorch)训练的分割结果与原始手稿(TensorFlow)报告的结果之间的差异。
Kumar数据集上的分割结果:
| 平台 | DICE | PQ | AJI |
|---|---|---|---|
| TensorFlow | 0.8258 | 0.5971 | 0.6412 |
| PyTorch | 0.8211 | 0.5904 | 0.6321 |
CoNSeP数据集上的分割结果:
| 平台 | DICE | PQ | AJI |
|---|---|---|---|
| TensorFlow | 0.8525 | 0.5477 | 0.5995 |
| PyTorch | 0.8504 | 0.5464 | 0.6009 |
可以在此处找到复现上述结果的检查点。
CoNSeP数据集上的同时分割和分类结果:
| 平台 | F1<sub>d</sub> | F1<sub>e</sub> | F1<sub>i</sub> | F1<sub>s</sub> | F1<sub>m</sub> |
|---|---|---|---|---|---|
| TensorFlow | 0.748 | 0.635 | 0.631 | 0.566 | 0.426 |
| PyTorch | 0.756 | 0.636 | 0.559 | 0.557 | 0.348 |
引用
如果使用了此代码的任何部分,请适当引用我们的论文。<br />
BibTex条目:<br />
@article{graham2019hover,
title={Hover-net: Simultaneous segmentation and classification of nuclei in multi-tissue histology images},
author={Graham, Simon and Vu, Quoc Dang and Raza, Shan E Ahmed and Azam, Ayesha and Tsang, Yee Wah and Kwak, Jin Tae and Rajpoot, Nasir},
journal={Medical Image Analysis},
pages={101563},
year={2019},
publisher={Elsevier}
}
作者
许可证
该项目根据MIT许可证授权 - 详见LICENSE文件。
请注意,PanNuke数据集根据Attribution-NonCommercial-ShareAlike 4.0 International许可,因此HoVer-Net的派生权重也根据相同许可共享。请考虑在该许可下使用权重对您的工作及其许可的影响。
编辑推荐精选


扣子-AI办公
职场AI,就用扣子
AI办公助手,复杂任务高效处理。办公效率低?扣子空间AI助手支持播客生成、PPT制作、网页开发及报告写作,覆盖科研、商业、舆情等领域的专家Agent 7x24小时响应,生活工作无缝切换,提升50%效率!


堆友
多风格AI绘画神器
堆友平台由阿里巴巴设计团队创建,作为一款AI驱动的设计工具,专为设计师提供一站式增长服务。功能覆盖海量3D素材、AI绘画、实时渲染以及专业抠图,显著提升设计品质��和效率。平台不仅提供工具,还是一个促进创意交流和个人发展的空间,界面友好,适合所有级别的设计师和创意工作者。


码上飞
零代码AI应用开发平台
零代码AI应用开发平台,用户只需一句话简单描述需求,AI能自动生成小程序、APP或H5网页应用,无需编写代码。

Vora
免费创建高清无水印Sora视频
Vora是一个免费创建高清无水印Sora视频的AI工具


Refly.AI
最适合小白的AI自动化工作流平台
无需编码,轻松生成可复用、可变现的AI自动化工作流


酷表ChatExcel
大模型驱动的Excel数据处理工具
基于大模型交互的表格处理系统,允许用户通过对话方式完成数据整理和可视化分析。系统采用机器学习算法解析用户指令,自动执行排序、公式计算和数据透视等操作,支持多种文件格式导入导出。数据处理响应速度保持在0.8秒以内,支持超过100万行数据的即时分析。


TRAE编程
AI辅助编程,代码自动修复
Trae是一种自适应的集成开发环境(IDE),通过自动化和多元协作改变开发流程。利用Trae,团队能够更快速、精确地编写和部署代码,从而提高编程效率和项目交付速度。Trae具备上下文感知和代码自动完成功能,是提升开发效率的理想工具。


AIWritePaper论文写作
AI论文写作指导平台
AIWritePaper论文写作是一站式AI论文写作辅助工具,简化了选题、文献检索至论文撰写的整个过程。通过简单设定,平台可快速生成高质量论文大纲和全文,配合图表、参考文献等一应俱全,同时提供开题报告和答辩PPT等增值服务,保障数据安全,有效提升写作效率和论文质量。

博思AIPPT
AI一键生成PPT,就用博思AIPPT!
博思AIPPT,新一代的AI生成PPT平台,支持智能生成PPT、AI美化PPT、文本&链接生成PPT、导入Word/PDF/Markdown文档生成PPT等,内置海量精美PPT模板,涵盖商务、教育、科技等不同风格,同时针对每个页面提供多种版式,一键自适应切换,完美适配各种办公场景。


潮际好麦
AI赋能电商视觉革命,一站式智能商拍平台
潮际好麦深耕服装行业,是国内AI试衣效果最好的软件。使用先进AIGC能力为电商卖家批量提供优质的、低成本的商拍图。合作品牌有Shein、Lazada、安踏、百丽等65个国内外头部品牌,以及国内10万+淘宝、天猫、京东等主流平台的品牌商家,为卖家节省将近85%的出图成本,提升约3倍出图效率,让品牌能够快速上架。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号





