RNA-FM
2024年3月更新:
- RNA家族聚类和RNA类型分类教程和教程视频(中文)。
- mRNA-FM,一个在mRNA中的编码序列(CDS)上预训练的基础模型现已发布!该模型可以接收CDS并用上下文嵌入表示它们,有利于mRNA和蛋白质相关任务。
本仓库包含RNA基础模型(RNA-FM)的代码和预训练模型。 RNA-FM在各种结构预测任务以及几个功能相关任务中的表现优于所有测试的单序列RNA语言模型。 您可以在我们的论文《Interpretable RNA Foundation Model from Unannotated Data for Highly Accurate RNA Structure and Function Predictions》(Chen等,2022年)中找到有关RNA-FM的更多详细信息。
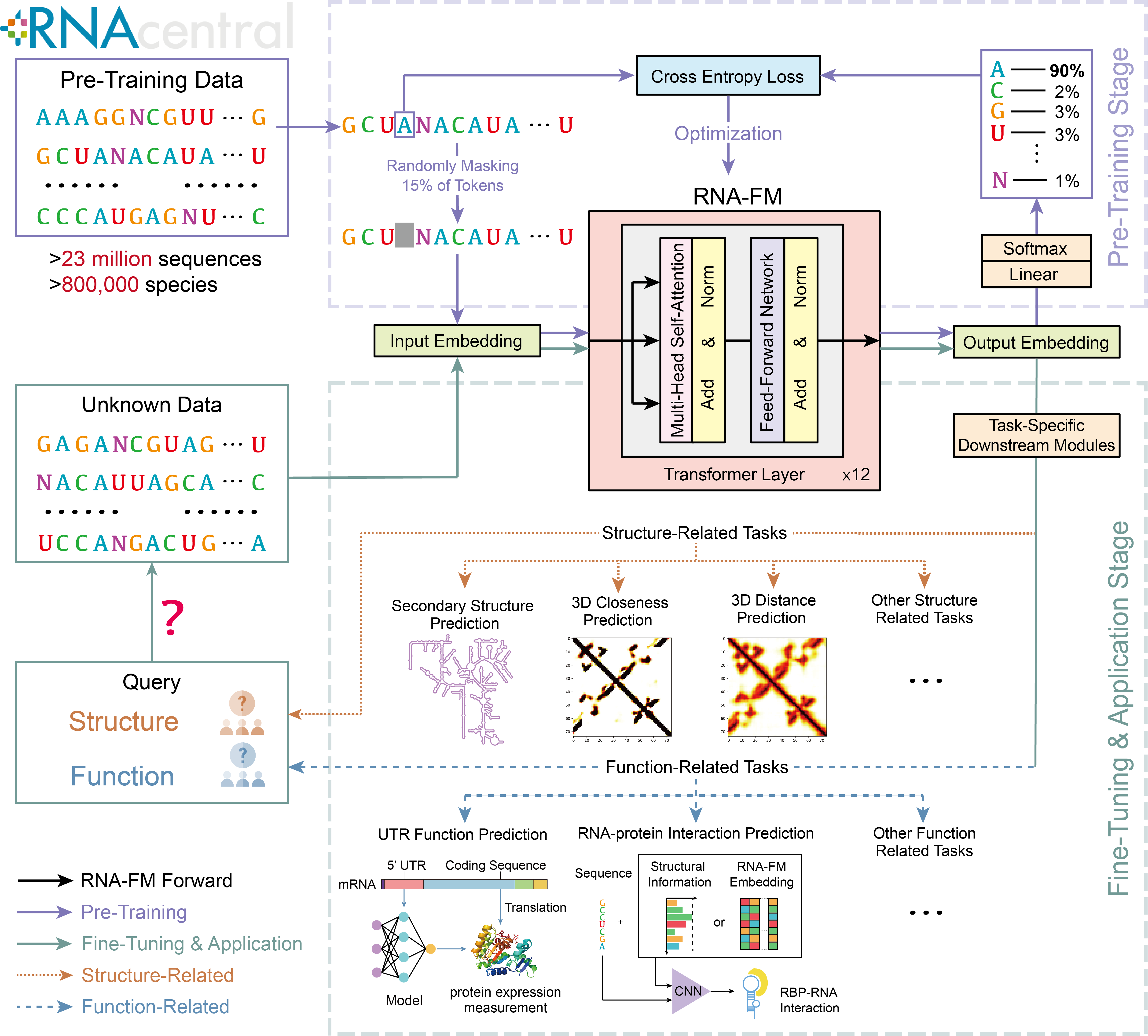
</details> <details><summary>目录</summary> </details>@article{chen2022interpretable, title={Interpretable rna foundation model from unannotated data for highly accurate rna structure and function predictions}, author={Chen, Jiayang and Hu, Zhihang and Sun, Siqi and Tan, Qingxiong and Wang, Yixuan and Yu, Qinze and Zong, Licheng and Hong, Liang and Xiao, Jin and King, Irwin and others}, journal={arXiv preprint arXiv:2204.00300}, year={2022} }
使用Conda创建环境 <a name="Setup_Environment"></a>
首先,下载仓库并创建环境。
git clone https://github.com/ml4bio/RNA-FM.git
cd ./RNA-FM
conda env create -f environment.yml
然后,激活"RNA-FM"环境并进入工作空间。
conda activate RNA-FM
cd ./redevelop
访问预训练模型。 <a name="Available_Pretrained_Models"></a>
从这个谷歌云端硬盘链接下载预训练模型,并将pth文件放入pretrained文件夹。
使用现有脚本应�用RNA-FM。 <a name="Usage"></a>
1. 嵌入提取。 <a name="RNA-FM_Embedding_Generation"></a>
python launch/predict.py --config="pretrained/extract_embedding.yml" \
--data_path="./data/examples/example.fasta" --save_dir="./resuts" \
--save_frequency 1 --save_embeddings
形状为(L,640)的RNA-FM嵌入将保存在$save_dir/representations中。
对于mRNA-FM,您可以使用额外的参数MODEL.BACKBONE_NAME来调用它:
python launch/predict.py --config="pretrained/extract_embedding.yml" \
--data_path="./data/examples/example.fasta" --save_dir="./resuts" \
--save_frequency 1 --save_embeddings --save_embeddings_format raw MODEL.BACKBONE_NAME mrna-fm
2. 下游预测 - RNA二级结构。 <a name="RNA_Secondary_Structure_Prediction"></a>
python launch/predict.py --config="pretrained/ss_prediction.yml" \
--data_path="./data/examples/example.fasta" --save_dir="./resuts" \
--save_frequency 1
预测的概率图将以.npy文件的形式保存,后处理的二元预测将以.ct文件的形式保存。您可以在$save_dir/r-ss中找到它们。
3. 在线版本 - RNA-FM服务器。 <a name="Server"></a>
如果您在部署RNA-FM的本地版本时遇到任何问题,可以通过这个链接访问其在线版本,RNA-FM服务器。 您可以轻松地在服务器上提交作业并随后下载结果,无需设置环境和占用任何计算资源。
进一步开发的快速入门。 <a name="Quick_Start"></a>
Python 3.8(可能更高版本)和PyTorch是使用此仓库必须安装的先决包。
如果您只想使用预训练的语言模型,可以使用以下pip命令在自己的环境中安装rna-fm。
您可以从PIPY安装rna-fm:
pip install rna-fm
或者从github安装rna-fm:
cd ./RNA-FM
pip install .
安装后,您可以使用以下代码加载RNA-FM并提取其嵌入:
import torch
import fm
# 加载RNA-FM模型
model, alphabet = fm.pretrained.rna_fm_t12()
batch_converter = alphabet.get_batch_converter()
model.eval() # 禁用dropout以获得确定性结果
# 准备数据
data = [
("RNA1", "GGGUGCGAUCAUACCAGCACUAAUGCCCUCCUGGGAAGUCCUCGUGUUGCACCCCU"),
("RNA2", "GGGUGUCGCUCAGUUGGUAGAGUGCUUGCCUGGCAUGCAAGAAACCUUGGUUCAAUCCCCAGCACUGCA"),
("RNA3", "CGAUUCNCGUUCCC--CCGCCUCCA"),
]
batch_labels, batch_strs, batch_tokens = batch_converter(data)
# 提取嵌入(在CPU上)
with torch.no_grad():
results = model(batch_tokens, repr_layers=[12])
token_embeddings = results["representations"][12]
更多教程可以在https://ml4bio.github.io/RNA-FM/找到。相关的笔记本存储在tutorials文件夹中。
对于mRNA-FM,上述代码需要稍作修改。需要注意的是,输入RNA序列的长度应为3的倍数,以确保序列可以被标记为一系列密码子(3-mer)。
import torch
import fm
# 加载mRNA-FM模型
model, alphabet = fm.pretrained.mrna_fm_t12()
batch_converter = alphabet.get_batch_converter()
model.eval() # 禁用dropout以获得确定性结果
# 准备数据
data = [
("CDS1", "AUGGGGUGCGAUCAUACCAGCACUAAUGCCCUCCUGGGAAGUCCUCGUGUUGCACCCCUA"),
("CDS2", "AUGGGGUGUCGCUCAGUUGGUAGAGUGCUUGCCUGGCAUGCAAGAAACCUUGGUUCAAUCCCCAGCACUGCA"),
("CDS3", "AUGCGAUUCNCGUUCCC--CCGCCUCC"),
]
batch_labels, batch_strs, batch_tokens = batch_converter(data)
# 提取嵌入(在CPU上)
with torch.no_grad():
results = model(batch_tokens, repr_layers=[12])
token_embeddings = results["representations"][12]
相关RNA语言模型(BERT风格) <a name="Review"></a>
| 简称 | 代码 | 主题 | 层数 | 嵌入维度 | 最大长度 | 输入 | 标记 | 数据集 | 描述 | 年份 | 发布者 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| RNA-FM | 有 | 非编码RNA | 12 | 640 | 1024 | 序列 | 碱基 | RNAcentral 19 (2300万个样本) | 首个通用RNA语言模型 | 2022.04 | arxiv/bioRxiv |
| RNABERT | 有 | 非编码RNA | 6 | 120 | 440 | 序列 | 碱基 | RNAcentral (762370) 和 Rfam 14.3 数据集(使用部分多序列比对训练) | 专门用于结构对齐和聚类 | 2022.02 | NAR Genomics and Bioinformatics |
| UNI-RNA | 无 | RNA | 24 | 1280 | $\infty$ | 序列 | 碱基 | RNAcentral、nt 和 GWH (10亿) | 比RNA-FM规模更大、数据集更广的通用模型 | 2023.07 | bioRxiv |
| RNA-MSM | 有 | 非编码RNA | 12 | 768 | 1024 | 多序列比对 | 碱基 | Rfam 14.7 中的4069个RNA家族 | 直接利用多序列比对的进化信息的模型 | 2023.03 | NAR |
| SpliceBERT | 有 | 前体mRNA | 6 | 1024 | 512 | 序列 | 碱基 | 200万个前体信使RNA (pre-mRNA) | 专门用于前体mRNA剪接 | 2023.05 | bioRxiv |
| CodonBERT | 无 | mRNA CDS | 12 | 768 | 512*2 | 序列 | 密码子 (3mer) | NCBI的1000万个mRNA | 仅关注mRNA的CDS,不包括UTR | 2023.09 | bioRxiv |
| UTR-LM | 有 | mRNA 5'UTR | 6 | 128 | $\infty$ | 序列 | 碱基 | Ensembl、eGFP、mCherry和Cao的70万个5'UTR | 用于5'UTR和mRNA表达相关任务 | 2023.10 | bioRxiv |
| 3UTRBERT | 有 | mRNA 3'UTR | 12 | 768 | 512 | 序列 | k-mer | 20,362个3'UTR | 用于3'UTR介导的基因调控任务 | 2023.09 | bioRxiv |
| BigRNA | 无 | DNA | - | - | - | 序列 | - | 数千个与基因组匹配的数据集 | 组织特异性RNA表达、剪接、微RNA位点和RNA结合蛋白 | 2023.09 | bioRxiv |
引用 <a name="citations"></a>
如果您在研究中发现这些模型有用,我们希望您能引用相关论文:
对于RNA-FM:
@article{chen2022interpretable, title={Interpretable rna foundation model from unannotated data for highly accurate rna structure and function predictions}, author={Chen, Jiayang and Hu, Zhihang and Sun, Siqi and Tan, Qingxiong and Wang, Yixuan and Yu, Qinze and Zong, Licheng and Hong, Liang and Xiao, Jin and King, Irwin and others}, journal={arXiv preprint arXiv:2204.00300}, year={2022} }
本代码的模型基于esm序列建模框架构建。 我们使用fairseq序列建模框架来训练我们的RNA语言模型。 我们非常感谢这两项优秀的工作!
许可证 <a name="license"></a>
本源代码根据MIT许可证授权,该许可证可在本源代码树根目录中的LICENSE文件中找到。
编辑推荐精选


扣子-AI办公
职场AI,就用扣子
AI办公助手,复杂任务高效处理。办公效率低?扣子空间AI助手支持播客生成、PPT制作、网页开发及报告写作,覆盖科研、商业、舆情等领域的专家Agent 7x24小时响应,生活工作无缝切换,提升50%效率!


堆友
多风格AI绘画神器
堆友平台由阿里巴巴设计团队创建,作为一款AI驱动的设计工具,专为设计师提供一站式增长服务。功能覆盖海量3D素材、AI绘画、实时渲染以及专业抠图,显著提升设计品质和效率。平台不仅提供工具,还是一个促进创意交流和个人发展的空间,界面友好,适合所有级别的设计师和创意工作者。


码上飞
零代码AI应用开发平台
零代码AI应用开发平台,用户只需一句话简单描述需求,AI能自动生成小程序、APP或H5网页应用,无需编写代码。

Vora
免费创建高清无水印Sora视频
Vora是一个免费创建高清无水印Sora视频的AI工具


Refly.AI
最适合小白的AI自动化工作流平台
无需编码,轻松生成可复用、可变现的AI自动化工作流


酷表ChatExcel
大模型驱动的Excel数据处理工具
基于大模型交互的表格处理系统,允许用户通过对话方式完成数据整理和可视化分析。系统采用机器学习算法解析用户指令,自动执行排序、公式计算和数据透视等操作,支持多种文件格式导入导出。数据处理响应速度保持在0.8秒以内,支持超过100万行数据的即时分析。


TRAE编程
AI辅助编程,代码自动修复
Trae是一种自适应的集成开发环境(IDE),通过自动化和多元协作改变开发流程。利用Trae,团队能够更快速、精确地编写和部署代码,从而提高编程效率和项目交付速度。Trae具备上下文感知和代码自动完成功能,是提升开发效率的理想工具。


AIWritePaper论文写作
AI论文写作指导平台
AIWritePaper论文写作是一站式AI论文写作辅助工具,简化了选题、文献检索至论文撰写的整个过程。通过简单设定,平台可快速生成高质量论文大纲和全文,配合图表、参考文献等一应俱全,同时提供开题报告和答辩PPT等增值服务,保障数据安全,有效提升写作效率和论文质量。

博思AIPPT
AI一键生成PPT,就用博思AIPPT!
博思AIPPT,新一代的AI生成PPT平台,支持智能生成PPT、AI美化PPT、文本&链接生成PPT、导入Word/PDF/Markdown文档生成PPT等,内置海量精美PPT模板,涵盖商务、教育、科技等不同风格,同时针对每个页面提供多种版式,一键自适应切换,完美适配各种办公场景。


潮际好麦
AI赋能电商视觉革命,一站式智能商拍平台
潮际好麦深耕服装行业,是国内AI试衣效果最好的软件。使用先进AIGC能力为电商卖家批量提供优质的、低成本的�商拍图。合作品牌有Shein、Lazada、安踏、百丽等65个国内外头部品牌,以及国内10万+淘宝、天猫、京东等主流平台的品牌商家,为卖家节省将近85%的出图成本,提升约3倍出图效率,让品牌能够快速上架。
推荐工具精�选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号





