
DNABERT_2
多物种基因组理解基础模型
DNABERT-2是一个针对多物种基因组理解的高效基础模型。该模型在28个GUE基准任务中表现优异,采用BPE替代k-mer标记化,ALiBi代替位置嵌入,并整合多项技术提升效率。DNABERT-2为基因组分析提供了强大工具,可用于序列分类、元素识别和功能预测等多种任务。
DNABERT-2:多物种基因组的高效基础模型和基准
本仓库包含:
- DNABERT-2:多物种基因组的高效基础模型和基准的官方实现
- 基因组理解评估(GUE):一个包含28个数据集的全面基准,用于多物种基因组理解评估。
目录
更新 (2024/02/14)
我们发布了DNABERT-S,这是一个基于DNABERT-2的基础模型,专门设计用于生成DNA嵌入,可以自然地在嵌入空间中聚类和分离不同物种的基因组。如果您感兴趣,请查看这里。
1. 简介
DNABERT-2是一个在大规模多物种基因组上训练的基础模型,在GUE基准的28个任务上达到了最先进的性能。它用BPE替代了k-mer分词,用带线性偏置的注意力(ALiBi)替代了位置嵌入,并incorporat其他技术以提高DNABERT的效率和有效性。
2. 模型和数据
预训练模型可在Hugging Face上获取,名为zhihan1996/DNABERT-2-117M。Hugging Face ModelHub链接。直接下载链接。
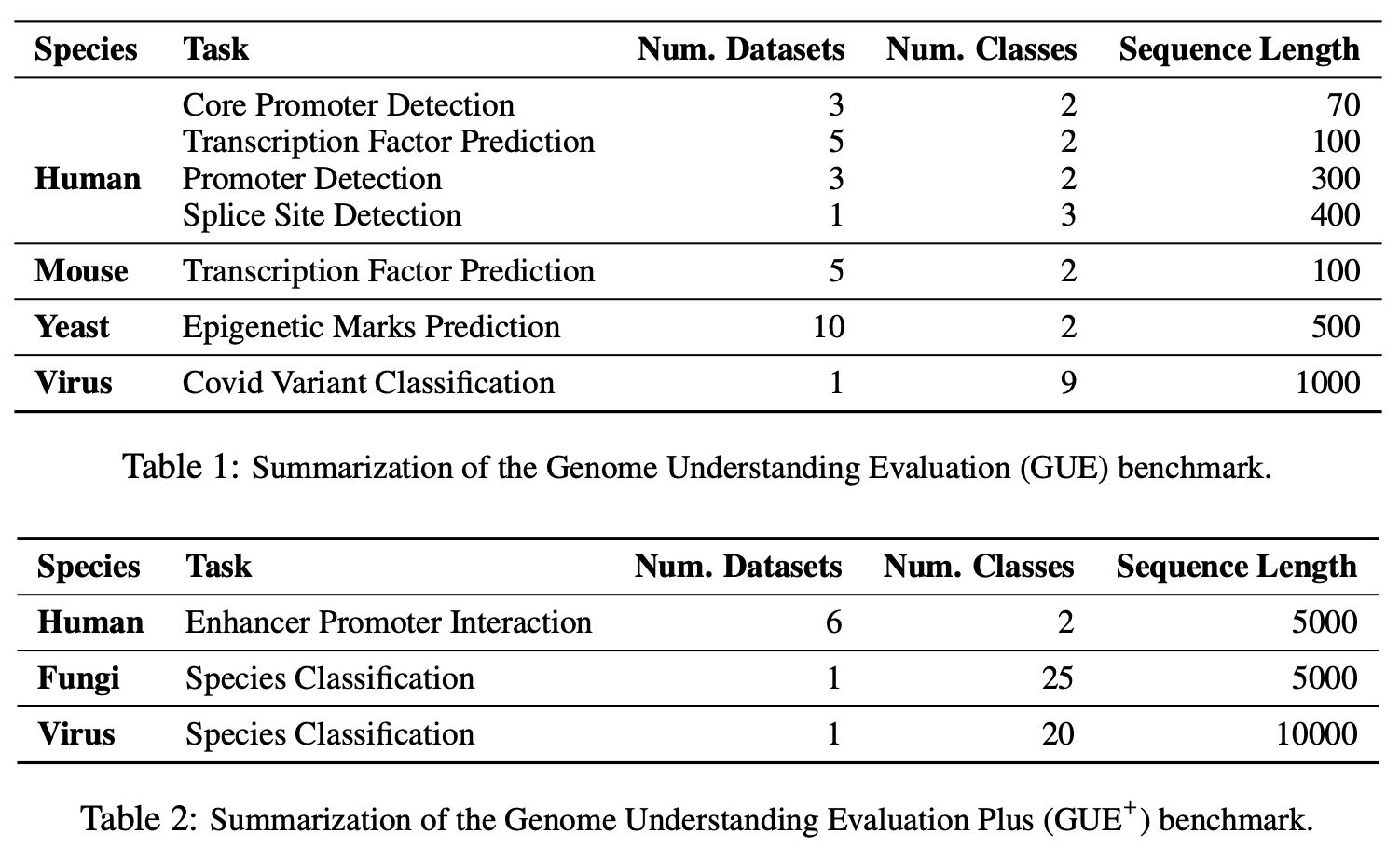
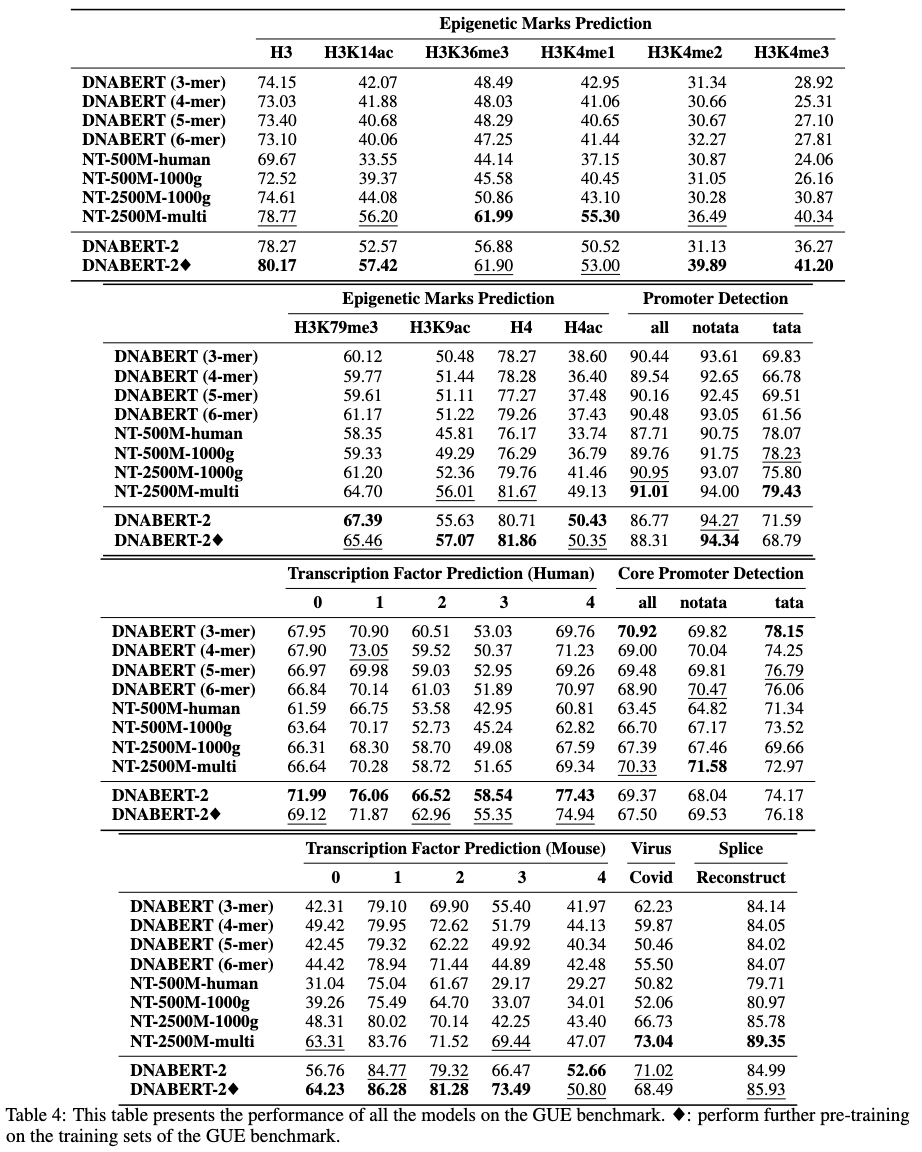
2.1 GUE:基因组理解评估
GUE是一个全面的基因组理解基准,由7个任务和4个物种的28个不同数据集组成。GUE可以在这里下载。GUE的统计数��据和模型性能如下所示:
3. 环境设置
# 创建并激活虚拟Python环境
conda create -n dna python=3.8
conda activate dna
# (如果您想使用flash attention,则为可选)
# 从源代码安装triton
git clone https://github.com/openai/triton.git;
cd triton/python;
pip install cmake; # 构建时依赖
pip install -e .
# 安装所需的包
python3 -m pip install -r requirements.txt
4. 快速开始
我们的模型可以通过transformers包轻松使用。
从Hugging Face加载模型(版本4.28):
import torch from transformers import AutoTokenizer, AutoModel tokenizer = AutoTokenizer.from_pretrained("zhihan1996/DNABERT-2-117M", trust_remote_code=True) model = AutoModel.from_pretrained("zhihan1996/DNABERT-2-117M", trust_remote_code=True)
从Hugging Face加载模型(版本 > 4.28):
from transformers.models.bert.configuration_bert import BertConfig config = BertConfig.from_pretrained("zhihan1996/DNABERT-2-117M") model = AutoModel.from_pretrained("zhihan1996/DNABERT-2-117M", trust_remote_code=True, config=config)
计算DNA序列的嵌入:
dna = "ACGTAGCATCGGATCTATCTATCGACACTTGGTTATCGATCTACGAGCATCTCGTTAGC"
inputs = tokenizer(dna, return_tensors = 'pt')["input_ids"]
hidden_states = model(inputs)[0] # [1, sequence_length, 768]
# 平均池化嵌入
embedding_mean = torch.mean(hidden_states[0], dim=0)
print(embedding_mean.shape) # 预期为768
# 最大池化嵌入
embedding_max = torch.max(hidden_states[0], dim=0)[0]
print(embedding_max.shape) # 预期为768
5. 预训练
我们使用并略微修改了MosaicBERT实现来训练DNABERT-2 https://github.com/mosaicml/examples/tree/main/examples/benchmarks/bert 。您应该能够按照说明复现模型训练。
或者,您可以使用 https://github.com/huggingface/transformers/tree/main/examples/pytorch/language-modeling 的run_mlm.py,通过从 https://huggingface.co/zhihan1996/DNABERT-2-117M/blob/main/bert_layers.py 导入BertModelForMaskedLM。这应该会产生一个非常相似的模型。
训练数据可在这里获取。
6. 微调
6.1 在GUE上评估模型
请先从这里下载GUE数据集。然后运行脚本以评估所有任务。
当前脚本设置为使用DataParallel在4个GPU上训练。如果您有不同数量的GPU,请相应地更改per_device_train_batch_size和gradient_accumulation_steps,以将全局批量大小调整为32,以复现论文中的结果。如果您想进行分布式多GPU训练(例如,��使用DistributedDataParallel),只需将python更改为torchrun --nproc_per_node ${n_gpu}。
export DATA_PATH=/path/to/GUE #(例如,/home/user)
cd finetune
# 在GUE上评估DNABERT-2
sh scripts/run_dnabert2.sh DATA_PATH
# 在GUE上评估DNABERT(例如,使用3-mer的DNABERT)
# 3表示3-mer,4表示4-mer,5表示5-mer,6表示6-mer
sh scripts/run_dnabert1.sh DATA_PATH 3
# 在GUE上评估Nucleotide Transformers
# 0表示500m-1000g,1表示500m-human-ref,2表示2.5b-1000g,3表示2.5b-multi-species
sh scripts/run_nt.sh DATA_PATH 0
6.2 在您自己的数据集上微调DNABERT2
这里我们提供了一个在您自己的数据集上微调DNABERT2的示例。
6.2.1 格式化您的数据集
首先,请从您的数据集生成3个csv文件:train.csv、dev.csv和test.csv。在训练过程中,模型会在train.csv上训练,并在dev.csv文件上进行评估。训练完成后,会加载在dev.csv文件上损失最小的检查点,并在test.csv上进行评估。如果您没有验证集,请将dev.csv和test.csv设置为相同的内容。
请查看sample_data文件夹以获取数据格式示例。每个文件应采用相同的格式,第一行为文档头,命名为sequence, label。每个后续行应包含一个DNA序列和一个数字标签,用逗号,连接(例如:ACGTCAGTCAGCGTACGT, 1)。
然后,您可以使用以下代码在自己的数据集上微调DNABERT-2:
cd finetune
export DATA_PATH=$path/to/data/folder # 例如,./sample_data
export MAX_LENGTH=100 # 请将此数值设置为您序列长度的0.25倍。
# 例如,如果您的DNA序列有1000个核苷酸碱基,则设置为250
# 这是因为分词器会将序列长度缩短约5倍
export LR=3e-5
# 使用DataParallel进行训练
python train.py \
--model_name_or_path zhihan1996/DNABERT-2-117M \
--data_path ${DATA_PATH} \
--kmer -1 \
--run_name DNABERT2_${DATA_PATH} \
--model_max_length ${MAX_LENGTH} \
--per_device_train_batch_size 8 \
--per_device_eval_batch_size 16 \
--gradient_accumulation_steps 1 \
--learning_rate ${LR} \
--num_train_epochs 5 \
--fp16 \
--save_steps 200 \
--output_dir output/dnabert2 \
--evaluation_strategy steps \
--eval_steps 200 \
--warmup_steps 50 \
--logging_steps 100 \
--overwrite_output_dir True \
--log_level info \
--find_unused_parameters False
# 使用DistributedDataParallel进行训练(更高效)
export num_gpu=4 # 请根据您的设置更改此值
torchrun --nproc-per-node=${num_gpu} train.py \
--model_name_or_path zhihan1996/DNABERT-2-117M \
--data_path ${DATA_PATH} \
--kmer -1 \
--run_name DNABERT2_${DATA_PATH} \
--model_max_length ${MAX_LENGTH} \
--per_device_train_batch_size 8 \
--per_device_eval_batch_size 16 \
--gradient_accumulation_steps 1 \
--learning_rate ${LR} \
--num_train_epochs 5 \
--fp16 \
--save_steps 200 \
--output_dir output/dnabert2 \
--evaluation_strategy steps \
--eval_steps 200 \
--warmup_steps 50 \
--logging_steps 100 \
--overwrite_output_dir True \
--log_level info \
--find_unused_parameters False
7. 引用
如果您对我们的论文或代码有任何疑问,请随时提出问题或发送电子邮件给Zhihan Zhou(zhihanzhou2020@u.northwestern.edu)。
如果您在工作中使用了DNABERT-2,请引用我们的论文:
DNABERT-2
@misc{zhou2023dnabert2,
title={DNABERT-2: Efficient Foundation Model and Benchmark For Multi-Species Genome},
author={Zhihan Zhou and Yanrong Ji and Weijian Li and Pratik Dutta and Ramana Davuluri and Han Liu},
year={2023},
eprint={2306.15006},
archivePrefix={arXiv},
primaryClass={q-bio.GN}
}
DNABERT
@article{ji2021dnabert,
author = {Ji, Yanrong and Zhou, Zhihan and Liu, Han and Davuluri, Ramana V},
title = "{DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome}",
journal = {Bioinformatics},
volume = {37},
number = {15},
pages = {2112-2120},
year = {2021},
month = {02},
issn = {1367-4803},
doi = {10.1093/bioinformatics/btab083},
url = {https://doi.org/10.1093/bioinformatics/btab083},
eprint = {https://academic.oup.com/bioinformatics/article-pdf/37/15/2112/50578892/btab083.pdf},
}
编辑推荐精选


扣子-AI办公
职场AI,就用扣子
AI办公助手,复杂任务高效处理。办公效率低?扣子空间AI助手支持播客生成、PPT制作、网页开发及报告写作,覆盖科研、商业、舆情等领域的专家Agent 7x24小时响应,生活工作无缝切换,提升50%效率!


堆友
多风格AI绘画神器
堆友平台由阿里巴巴设计团队创建,作为一款AI驱动的设计工具,专为设计师提供一站式增长服务。功能覆盖海量3D素材、AI绘画、实时渲染以及专业抠图,显著提升设计品质和效率。平台不仅提供工具,还是一个促进创意交流和个人发展的空间,界面友好,适合所有级别的设计师和创意工作者。


码上飞
零代码AI应用开发平台
零代码AI应用开发平台,用户只需一句话简单描述需求,AI能自动生成小程序、APP或H5网页应用,无需编写代码。

Vora
免费创建高清无水印Sora视频
Vora是�一个免费创建高清无水印Sora视频的AI工具


Refly.AI
最适合小白的AI自动化工作流平台
无需编码,轻松生成可复用、可变现的AI自动化工作流


酷表ChatExcel
大模型驱动的Excel数据处理工具
基于大模型交互的表格处理系统,允许用户通过对话方式完成数据整理和可视化分析。系统采用机器学习算法解析用户指令,自动执行排序、公式计算和数据透视等操作,支持多种文件格式导入导出。数据处理响应速度保持在0.8秒以内,支持超过100万行数据的即时分析。


TRAE编程
AI辅助编程,代码自动修复
Trae是一种自适应的集成开发环境(IDE),通过自动化和多元协作改变开发流程。利用Trae,团队能够更快速、精确地编写和部署代码,从而提高编程效率和项目交付速度。Trae具备上下文感知和代码自动完成功能,是提升开发效率的理想工具。


AIWritePaper论文写作
AI论文写作指导平台
AIWritePaper论文写作是一站式AI论文写作辅助工具,简化了选题、文献检索至论文撰写的整个过程。通过简单设定,平台可快速生成高质量论文大纲和全文,配合图表、参考文献等一应俱全,同时提供开题报告和答辩PPT等增值服务,保障数据安全,有效提升写作效率和论文质量。

博思AIPPT
AI一键生成PPT,就用博思AIPPT!
博思AIPPT,新一代的AI生成PPT平台,支持智能生成PPT、AI美化PPT、文本&链接生成PPT、导入Word/PDF/Markdown文档生成PPT等,内置海量精美PPT模板,涵盖商务、教育、科技等不同风格,同时针对每个页面提供多种版式,一键自适应切换,完美适配各种办公场景。


潮际好麦
AI赋能电商视觉革命,一站式智能商拍平台
潮际好麦深耕服装行业,是国内AI试衣效果最好的软件。使用先进AIGC能力为电商卖家批量提供优质的、低成本的商拍图。合作品牌有Shein、Lazada、安踏、百丽等65个国内外头部品牌,以及国内10万+淘宝、天猫、京东等主流平台的品牌商家,为卖家节省将近85%的出图成本,提升约3倍出图效率,让品牌能够快速上架。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号




