SysIdentPy提供最先进的技术来构建您的NARMAX模型,包括其变体NARX、NARMA、NAR、NFIR、ARMAX、ARX、ARMA等。它还包含大量有趣的示例,帮助您使用SysIdentPy构建非线性预测模型。
目录
简介
SysIdentPy是一个基于numpy构建的开源Python模块,用于使用NARMAX模型进行系统辨识,并在3-Clause BSD许可下分发。SysIdentPy为构建时间序列和动态系统的动态非线性模型提供了一个易于使用且灵活的框架。
使用SysIdentPy,您可以:
- 构建和自定义非线性预测模型。
- 使用最先进的技术进行模型结构选择和参数估计。
- 尝试神经NARX模型和其他先进算法。
查看我们的文档!
如需深入了解,请查看我们的配套书籍:
<a href="https://sysidentpy.org/book/0%20-%20Preface/"> <img src="https://yellow-cdn.veclightyear.com/0a4dffa0/4b5b332c-7198-4710-a22f-d1da4ba6906e.png" alt="非线性系统辨识" style="width: 200px; height: auto;" /> </a>如何安装SysIdentPy?
安装SysIdentPy最简单的方法是使用pip
pip install sysidentpy
要求
SysIdentPy需要:
- Python (>= 3.7)
- NumPy (>= 1.9.2) 用于数值算法
- Matplotlib >= 3.3.2 用于静态绘图和可视化
- Pytorch (>=1.7.1) 用于构建NARX神经网络
- scipy (>= 1.7.0) 用于数值和优化算法
该库兼容Linux、Windows和macOS。某些示例可能还需要额外的包,如pandas。
更多详情,请查看我们的安装指南
SysIdentPy的主要特性是什么?
| 特性 | 这是什么? |
|---|---|
| NARMAX理念 | 您可以构建NARMAX模型的变体,如NARX、NAR、NARMA、NFIR、ARMA、ARX、AR等。 |
| 模型结构选择 | 易于使用的方法来选择构建模型的最佳项,包括FROLS和MetaMSS,以及与参数估计技术的多种组合来选择模型项。 |
| 基函数 | 您可以使用不同的基函数来构建模型。您可以设置线性和非线性基函数,并将它们组合以获得自定义NARMAX模型。 |
| 参数估计 | 超过15种方法来估计模型参数并测试不同的结构选择场景。 |
| 多目标参数估计 | 您可以使用仿射信息来估计模型参数,最小化不同的目标函数。 |
| 模型仿真 | 您可以使用SimulateNARMAX类轻松重现论文中的结果。此外,您可以使用不同的参数估计方法测试已发表的模型并比较性能。 |
| 神经NARX | 您可以使用SysIdentPy与Pytorch一起创建自定义神经NARX模型架构,支持Pytorch的所有优化器和损失函数。 |
| 通用估计器 | 您可以使用来自scikit-learn、Catboost等包的估计器以及许多其他兼容接口和组合工具来创建NARMAX模型。 |
SysIdentPy 为何而生?
SysIdentPy 旨在成为一个免费开源的软件包,帮助社区为系统识别和时间序列预测设计 NARMAX 模型。更重要的是,它是目前最常用的 NARMAX 模型构建工具之一 —— Matlab 系统识别工具箱的免费且强大的替代品。
该项目由 Wilson R. L. Junior 积极维护,并正在寻找贡献者。
如何使用 SysIdentPy?
SysIdentPy 文档包含超过 20 个示例,帮助您入门:
- 典型的"Hello World"示例,入门级介绍 SysIdentPy 的主要概念
- 专门介绍 SysIdentPy 特性的章节��,如模型结构选择算法、基函数、参数估计等
- 专门介绍使用 SysIdentPy 处理真实世界数据集的用例章节。此外,还有与其他时间序列工具(如 Prophet、Neural Prophet、ARIMA 等)的简要比较和基准测试
示例
from torch import nn import numpy as np import pandas as pd import matplotlib.pyplot as plt from sysidentpy.metrics import root_relative_squared_error from sysidentpy.utils.generate_data import get_siso_data # 生成模拟动态系统的数据集 x_train, x_valid, y_train, y_valid = get_siso_data( n=1000, colored_noise=False, sigma=0.001, train_percentage=80 )
使用 FROLS 算法构建多项式 NARX 模型
from sysidentpy.model_structure_selection import FROLS from sysidentpy.basis_function import Polynomial from sysidentpy.utils.display_results import results from sysidentpy.utils.plotting import plot_residues_correlation, plot_results from sysidentpy.residues.residues_correlation import compute_residues_autocorrelation from sysidentpy.residues.residues_correlation import compute_cross_correlation basis_function=Polynomial(degree=2) model = FROLS( order_selection=True, n_info_values=10, extended_least_squares=False, ylag=2, xlag=2, info_criteria='aic', estimator='least_squares', basis_function=basis_function ) model.fit(X=x_train, y=y_train) yhat = model.predict(X=x_valid, y=y_valid) rrse = root_relative_squared_error(y_valid, yhat) print(rrse) r = pd.DataFrame( results( model.final_model, model.theta, model.err, model.n_terms, err_precision=8, dtype='sci' ), columns=['回归量', '参数', 'ERR']) print(r) 回归量 参数 ERR 0 x1(k-2) 0.9000 0.95556574 1 y(k-1) 0.1999 0.04107943 2 x1(k-1)y(k-1) 0.1000 0.00335113 plot_results(y=y_valid, yhat=yhat, n=1000) ee = compute_residues_autocorrelation(y_valid, yhat) plot_residues_correlation(data=ee, title="残差", ylabel="$e^2$") x1e = compute_cross_correlation(y_valid, yhat, x2_val) plot_residues_correlation(data=x1e, title="残差", ylabel="$x_1e$")

NARX 神经网络
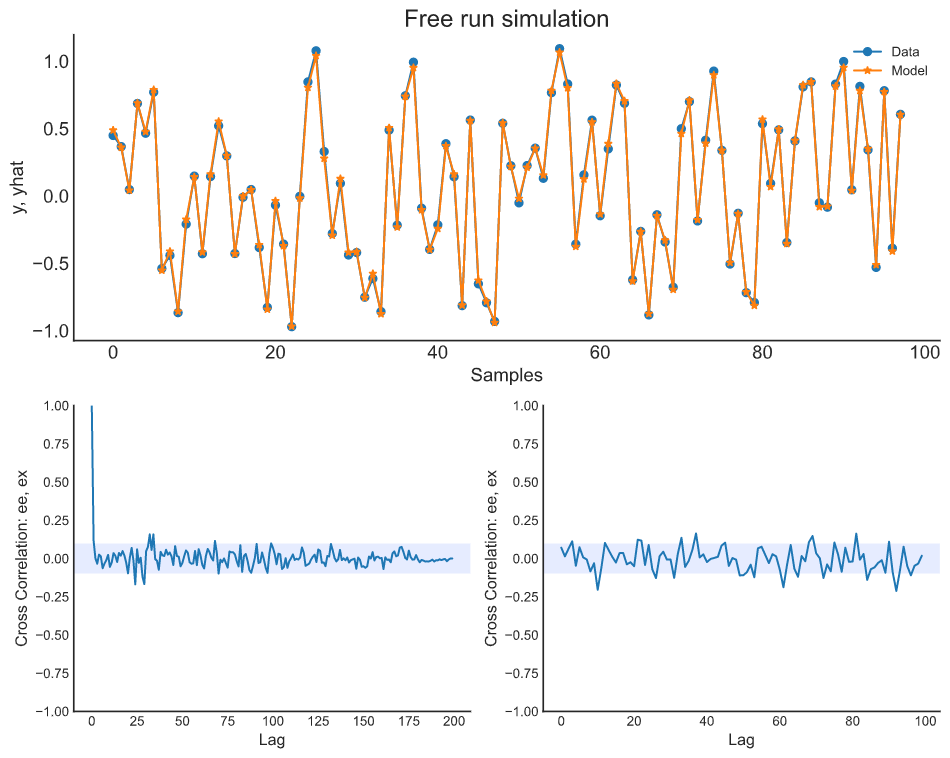
from sysidentpy.neural_network import NARXNN from sysidentpy.basis_function import Polynomial from sysidentpy.utils.plotting import plot_residues_correlation, plot_results from sysidentpy.residues.residues_correlation import compute_residues_autocorrelation from sysidentpy.residues.residues_correlation import compute_cross_correlation class NARX(nn.Module): def __init__(self): super().__init__() self.lin = nn.Linear(4, 10) self.lin2 = nn.Linear(10, 10) self.lin3 = nn.Linear(10, 1) self.tanh = nn.Tanh() def forward(self, xb): z = self.lin(xb) z = self.tanh(z) z = self.lin2(z) z = self.tanh(z) z = self.lin3(z) return z basis_function=Polynomial(degree=1) narx_net = NARXNN( net=NARX(), ylag=2, xlag=2, basis_function=basis_function, model_type="NARMAX", loss_func='mse_loss', optimizer='Adam', epochs=200, verbose=False, optim_params={'betas': (0.9, 0.999), 'eps': 1e-05} # 优化器的可选参数 ) narx_net.fit(X=x_train, y=y_train) yhat = narx_net.predict(X=x_valid, y=y_valid) plot_results(y=y_valid, yhat=yhat, n=1000) ee = compute_residues_autocorrelation(y_valid, yhat) plot_residues_correlation(data=ee, title="残差", ylabel="$e^2$") x1e = compute_cross_correlation(y_valid, yhat, x_valid) plot_residues_correlation(data=x1e, title="残差", ylabel="$x_1e$")
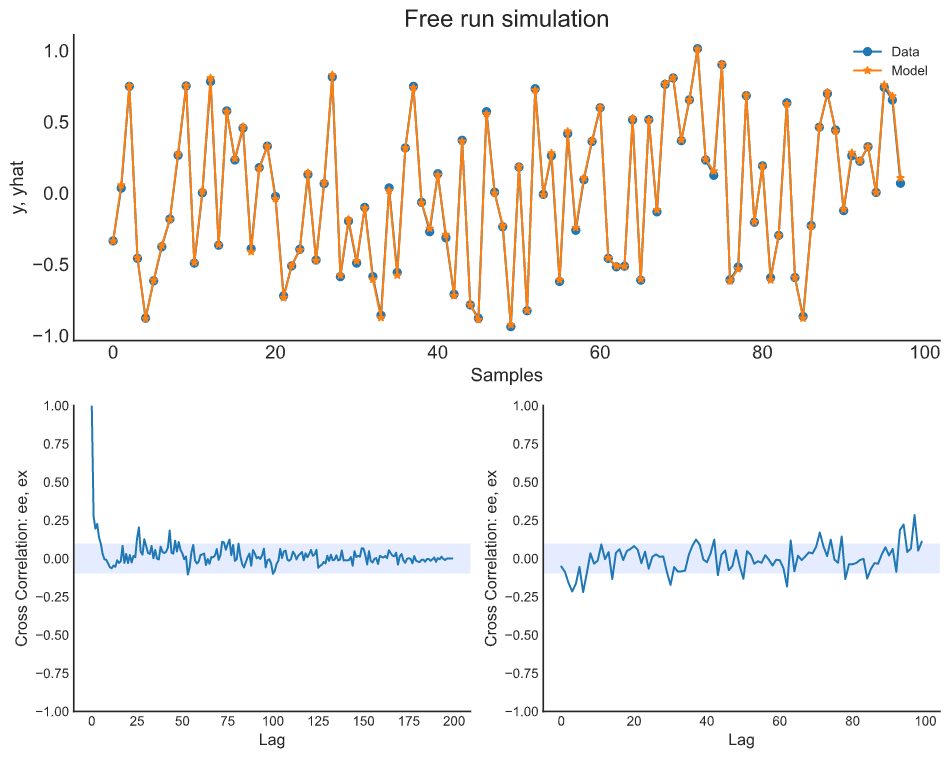
Catboost-narx
from catboost import CatBoostRegressor from sysidentpy.general_estimators import NARX from sysidentpy.basis_function import Polynomial from sysidentpy.utils.plotting import plot_residues_correlation, plot_results from sysidentpy.residues.residues_correlation import compute_residues_autocorrelation from sysidentpy.residues.residues_correlation import compute_cross_correlation basis_function=Polynomial(degree=1) catboost_narx = NARX( base_estimator=CatBoostRegressor( iterations=300, learning_rate=0.1, depth=6), xlag=2, ylag=2, basis_function=basis_function, fit_params={'verbose': False} ) catboost_narx.fit(X=x_train, y=y_train) yhat = catboost_narx.predict(X=x_valid, y=y_valid) plot_results(y=y_valid, yhat=yhat, n=1000) ee = compute_residues_autocorrelation(y_valid, yhat) plot_residues_correlation(data=ee, title="残差", ylabel="$e^2$") x1e = compute_cross_correlation(y_valid, yhat, x_valid) plot_residues_correlation(data=x1e, title="残差", ylabel="$x_1e$")
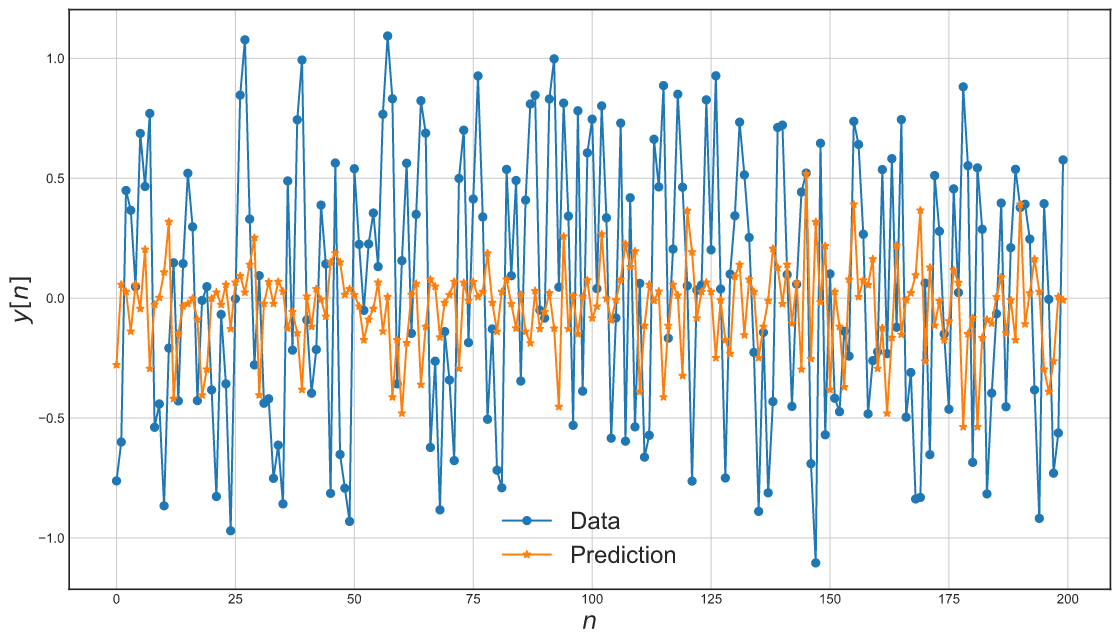
不使用NARX配置的Catboost
以下是不使用NARX配置的Catboost性能。
def plot_results_tmp(y_valid, yhat): _, ax = plt.subplots(figsize=(14, 8)) ax.plot(y_valid[:200], label='数据', marker='o') ax.plot(yhat[:200], label='预测', marker='*') ax.set_xlabel("$n$", fontsize=18) ax.set_ylabel("$y[n]$", fontsize=18) ax.grid() ax.legend(fontsize=18) plt.show() catboost = CatBoostRegressor( iterations=300, learning_rate=0.1, depth=6 ) catboost.fit(x_train, y_train, verbose=False) plot_results_tmp(y_valid, catboost.predict(x_valid))
examples目录中有几个Jupyter笔记本,其中包含如何使用该软件包的教程以及sysidentpy的一些具体应用。试试看吧!
交流
-
Discord服务器:https://discord.gg/8eGE3PQ
引用
��如果您在项目中使用了SysIdentPy,请给我发邮件。
如果您在科学出版物中使用了SysIdentPy,我们将感谢您引用以下论文:
- Lacerda et al., (2020). SysIdentPy: A Python package for System Identification using NARMAX models. Journal of Open Source Software, 5(54), 2384, https://doi.org/10.21105/joss.02384
@article{Lacerda2020,
doi = {10.21105/joss.02384},
url = {https://doi.org/10.21105/joss.02384},
year = {2020},
publisher = {The Open Journal},
volume = {5},
number = {54},
pages = {2384},
author = {Wilson Rocha Lacerda Junior and Luan Pascoal Costa da Andrade and Samuel Carlos Pessoa Oliveira and Samir Angelo Milani Martins},
title = {SysIdentPy: A Python package for System Identification using NARMAX models},
journal = {Journal of Open Source Software}
}
灵感来源
文档和结构(甚至包括这一部分)公开地受到Scikit-learn、EinsteinPy和许多其他项目的启发,因为我们一直在使用它们来学习。
贡献者
<a href="https://github.com/wilsonrljr/sysidentpy/graphs/contributors"> <img src="https://contributors-img.web.app/image?repo=wilsonrljr/sysidentpy" width = 500/> </a>赞助商
<!-- sponsors -->特别感谢我们的赞助商
<hr />黄金赞助商
<a href="https://github.com/ml-technium"> <img alt="Technium" src="https://avatars.githubusercontent.com/u/83768197?v=4" width="90" height="90"> </a>月度赞助商
<a href="https://github.com/statisticallyinsifnificant"> <img alt="statisticallyinsifnificant" src="https://avatars.githubusercontent.com/u/158107107?v=4" width="90" height="90"> </a>个人赞助商
<a href="https://github.com/nataliakeles"> <img alt="Nath Keles" src="https://avatars.githubusercontent.com/u/61664158?v=4" width="90" height="90"> </a> <hr /> <!-- sponsors -->编辑推荐精选


扣子-AI办公
职场AI,就用扣子
AI办公助手,复杂任务高效处理。办公效率低?扣子��空间AI助手支持播客生成、PPT制作、网页开发及报告写作,覆盖科研、商业、舆情等领域的专家Agent 7x24小时响应,生活工作无缝切换,提升50%效率!


堆友
多风格AI绘画神器
堆友平台由阿里巴巴设计团队创建,作为一款AI驱动的设计工具,专为设计师提供一站式增长服务。功能覆盖海量3D素材、AI绘画、实时渲染以及专业抠图,显著提升设计品质和效率。平台不仅提供工具,还是一个促进创意交流和个人发展的空间,界面友好,适合所有级别的设计师和创意工作者。


码上飞
零代码AI应用开发平台
零代码AI应用开发平台,用户只需一句话简单描述需求,AI能自动生成小程序、APP或H5网页应用,无需编写代码。

Vora
免费创建高清无水印Sora视频
Vora是一个免费创建高清无水印Sora视频的AI工具


Refly.AI
最适合小白的AI自动化工作流平台
无需编码,轻松生成可复用、可变现的AI自动化工作流


酷表ChatExcel
大模型驱动的Excel数据处理工具
基于大模型交互的表格处理系统,允许用户通过对话方式完成数据整理和可视化分析。系统采用机器学习算法解析用户指令,自动执行排序、公式计算和数据透视等操作,支持多种文件格式导入导出。数据处理响应速度保持在0.8秒以内,支持超过100万行数据的即时分析。


TRAE编程
AI辅助编程,代码自动修复
Trae是一种自适应的集成开发环境(IDE),通过自动化和多元协作改变开发流程。利用Trae,团队能够更快速、精确地编写和部署代码,从而提高编程效率和项目交付速度。Trae具备上下文感知和代码自动完成功能,是提升开发效率的理想��工具。


AIWritePaper论文写作
AI论文写作指导平台
AIWritePaper论文写作是一站式AI论文写作辅助工具,简化了选题、文献检索至论文撰写的��整个过程。通过简单设定,平台可快速生成高质量论文大纲和全文,配合图表、参考文献等一应俱全,同时提供开题报告和答辩PPT等增值服务,保障数据安全,有效提升写作效率和论文质量。

博思AIPPT
AI一键生成PPT,就用博思AIPPT!
博思AIPPT,新一代的AI生成PPT平台,支持智能生成PPT、AI美化PPT、文本&链接生成PPT、导入Word/PDF/Markdown文档生成PPT等,内置海量精美PPT模板,涵盖商务、教育、科技等不同风格,同时针对每个页面提供多种版式,一键自适应切换,完美适配各种办公场景。


潮际好麦
AI赋能电商视觉革命,一站式智能商拍平台
潮际好麦深耕服装行业,是国内AI试衣效果最好的软件。使用先进AIGC能力为电商卖家批量提供优质的、低成本的商拍图。合作品牌有Shein、Lazada、安踏、百丽等65个国内外头部品牌,以及国内10万+淘宝、天猫、京东等主流平台的品牌商家,为卖家节省将近85%的出图成本,提升约3倍出图效率,让品牌能够快速上架。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号
















