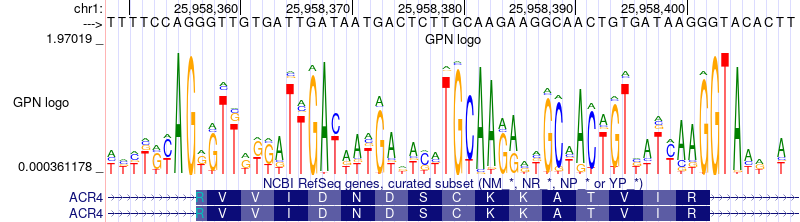
GPN (基因组预训练网络)
[ ](https://raw.githubusercontent.com/songlab-cal/gpn/main/ https://genome.ucsc.edu/s/gbenegas/gpn-arabidopsis)
](https://raw.githubusercontent.com/songlab-cal/gpn/main/ https://genome.ucsc.edu/s/gbenegas/gpn-arabidopsis)
目录
安装
pip install git+https://github.com/songlab-cal/gpn.git
最小使用示例
import gpn.model from transformers import AutoModelForMaskedLM model = AutoModelForMaskedLM.from_pretrained("songlab/gpn-brassicales") # 或 model = AutoModelForMaskedLM.from_pretrained("songlab/gpn-msa-sapiens")
GPN
也可称为GPN-SS(单序列)。
示例
特定论文的代码和资源
使用自己的数据进行训练
- [创建数据集的Snakemake工作流程](https://github.com/songlab-cal/gpn/blob/main/workflow/make_dataset
- 可以根据给定的登录号列表自动从NCBI下载数据,或使用您自己的fasta文件。
- 训练
- 将自动检测所有可用的GPU。
- 在Weights & Biases上跟踪指标。
- 已实现的模型:
ConvNet,GPNRoFormer(Transformer) - 指定配置覆盖:例如
--config_overrides n_layers=30 - 示例:
WANDB_PROJECT=your_project torchrun --nproc_per_node=$(echo $CUDA_VISIBLE_DEVICES | awk -F',' '{print NF}') -m gpn.ss.run_mlm --do_train --do_eval \ --fp16 --report_to wandb --prediction_loss_only True --remove_unused_columns False \ --dataset_name results/dataset --tokenizer_name gonzalobenegas/tokenizer-dna-mlm \ --soft_masked_loss_weight_train 0.1 --soft_masked_loss_weight_evaluation 0.0 \ --weight_decay 0.01 --optim adamw_torch \ --dataloader_num_workers 16 --seed 42 \ --save_strategy steps --save_steps 10000 --evaluation_strategy steps \ --eval_steps 10000 --logging_steps 10000 --max_steps 120000 --warmup_steps 1000 \ --learning_rate 1e-3 --lr_scheduler_type constant_with_warmup \ --run_name your_run --output_dir your_output_dir --model_type ConvNet \ --per_device_train_batch_size 512 --per_device_eval_batch_size 512 --gradient_accumulation_steps 1 \ --torch_compile
- 提取嵌入
- 输入文件需要包含
chrom、start、end - 示例:
- 输入文件需要包含
torchrun --nproc_per_node=$(echo $CUDA_VISIBLE_DEVICES | awk -F',' '{print NF}') -m gpn.ss.get_embeddings windows.parquet genome.fa.gz 100 your_output_dir \ results.parquet --per-device-batch-size 4000 --is-file --dataloader-num-workers 16
- 变异效应预测
- 输入文件需要包含
chrom、pos、ref、alt - 示例:
- 输入文件需要包含
torchrun --nproc_per_node=$(echo $CUDA_VISIBLE_DEVICES | awk -F',' '{print NF}') -m gpn.ss.run_vep variants.parquet genome.fa.gz 512 your_output_dir results.parquet \ --per-device-batch-size 4000 --is-file --dataloader-num-workers 16
GPN-MSA
示例
- 模型使用示例:
examples/msa/basic_example.ipynb - 变异效应预测:
examples/msa/vep.ipynb - 训练(人类):
examples/msa/training.ipynb
特定论文的代码和资源
在其他物种上训练(如植物)
正在建设中。
引用
GPN:
@article{benegas2023dna, author = {Gonzalo Benegas and Sanjit Singh Batra and Yun S. Song }, title = {DNA language models are powerful predictors of genome-wide variant effects}, journal = {Proceedings of the National Academy of Sciences}, volume = {120}, number = {44}, pages = {e2311219120}, year = {2023}, doi = {10.1073/pnas.2311219120}, URL = {https://www.pnas.org/doi/abs/10.1073/pnas.2311219120}, eprint = {https://www.pnas.org/doi/pdf/10.1073/pnas.2311219120}, }
GPN-MSA:
@article{benegas2023gpnmsa, author = {Gonzalo Benegas and Carlos Albors and Alan J. Aw and Chengzhong Ye and Yun S. Song}, title = {GPN-MSA: an alignment-based DNA language model for genome-wide variant effect prediction}, elocation-id = {2023.10.10.561776}, year = {2023}, doi = {10.1101/2023.10.10.561776}, publisher = {Cold Spring Harbor Laboratory}, URL = {https://www.biorxiv.org/content/early/2023/10/11/2023.10.10.561776}, eprint = {https://www.biorxiv.org/content/early/2023/10/11/2023.10.10.561776.full.pdf}, journal = {bioRxiv} }
编辑推荐精选


扣子-AI办公
职场AI,就用扣子
AI办公助手,复杂任务高效处理。办公效率低?扣子空间AI助手支持播客生成、PPT制作、网页开发及报告写作,覆盖科研、商业、舆情等领域的专家Agent 7x24小时响应,生活工作无缝切换,提升50%效率!


堆友
多风格AI绘画神器
堆友平台由阿里巴巴设计团队创建,作为一款AI驱动的设计工具,专为设计师提供一站式增长服务。功能覆盖海量3D素材、AI绘画、实时渲染以及专业抠图,显著提升设计品质和效率。平台不仅提供工具,还是一个促进创意交流和个人发展的空间,界面友好,适合所有级别的设计师和创意工作者。


码上飞
零代码AI应用开发平台
零代码AI应用开发平台,用户只需一句话简单描述需求,AI能自动生成小��程序、APP或H5网页应用,无需编写代码。

Vora
免费创建高清无水印Sora视频
Vora是一个免费创建高清无水印Sora视频的AI工具


Refly.AI
最适合小白的AI自动化工作流平台
无需编码,轻松生成可复用、可变现的AI自动化工作流


酷表ChatExcel
大模型驱动的Excel数据处理工具
基于大模型交互的表格处理系统,允许用户通过对话方式完成数据整理和可视化分析。系统采用机器学习算法解析用户指令,自动执行排序、公式计算和数据透视等操作,支持多种文件格式导入导出。数据处理响应速度保持在0.8秒以内,支持超过100万行数据的即时分析。


TRAE编程
AI辅助编程,代码自动修复
Trae是一种自适应的集成开发环境(IDE),通过自动化和多元协作改变开发流程。利用Trae,团队能够更快速、精确地编写和部署代码,从而提高编程效率和项目交付速度。Trae具备上下文感知和代码自动完成功能,是提升开发效率的理想工具。


AIWritePaper论文写作
AI论文写作指导平台
AIWritePaper论文写作是一站式AI论文写作辅助工具,简化了选题、文献检索至论文撰写的整个过程。通过简单设定,平台可快速生成高质量论文大纲和全文,配合图表、参考文献等一应俱全,同时提供开题报告和答辩PPT等增值服务,保障数据安全,有效提升写作效率和论文质量。

博思AIPPT
AI一键生成PPT,就用博思AIPPT!
博思AIPPT,新一代的AI生成PPT平台,支持智能生成PPT、AI美化PPT、文本&链接生成PPT、导入Word/PDF/Markdown文档生成PPT等,内置海量精美PPT模板,涵盖商务、教育、科技等不同风格,同时针对每个页面提供多种版式,一键自适应切换,完美适配各种办公场景。


潮际好麦
AI赋能电商视觉革命,一站式智能商拍平台
潮际好麦深耕服装行业,是国内AI试衣效果最好的软件。使用先进AIGC能力为电商卖家批量提供优质的、低成本的商拍图。合作品牌有Shein、Lazada、安踏、百丽等65个国内外头部品牌,以及国内10万+淘宝、天猫、京东等主流平台的品牌商家,为卖家节省将近85%的出图成本,提升约3倍出图效率,让品牌能够快速上架。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号





